原标题:哈佛大学的基因编辑工具选择将DNA“潜入”细胞而不进行切割 来源:cnBeta.COM
CRISPR-Cas9是一种革命性的基因编辑工具,但它并非没有缺点。现在,哈佛大学的科学家们展示了一种被称为Retron Library Recombineering(RLR)的替代性基因工程系统,它在不切割DNA的情况下工作,可以快速应用于巨大的细胞群。
CRISPR的工作方式就像一把基因剪刀,能够对活细胞的基因组进行精确的剪切和粘贴编辑。该系统可以寻找到一个特定的DNA序列,然后使用一种酶,最常见的是Cas9来进行切割。当细胞执行其DNA修复程序时,CRISPR指示它使用不同的序列而不是原来的序列,从而实现编辑基因组的目的。
这个系统已经被证明在一系列的应用中是非常有价值的,从治疗癌症、艾滋病和肌肉萎缩症等疾病,到害虫控制、改善农作物,以及用细菌制造生物计算机。
然而,也有潜在的问题。切割DNA可能会导致一些意想不到的副作用,而且有人担心CRISPR可能会在基因组的错误部分进行编辑。对于大规模修剪工作来说,CRISPR也可能是一个有点棘手的问题,因为很难跟踪突变体在实验室测试中产生了哪些影响。
哈佛医学院和怀斯研究所的研究人员提出的新基因编辑技术试图解决这些问题。RLR的主要区别在于它根本不切割DNA--相反,它在细胞分裂前复制其基因组时引入了新的DNA片段。
一张示意图说明了新的Retron Library Recombineering(RLR)基因编辑技术如何运作:
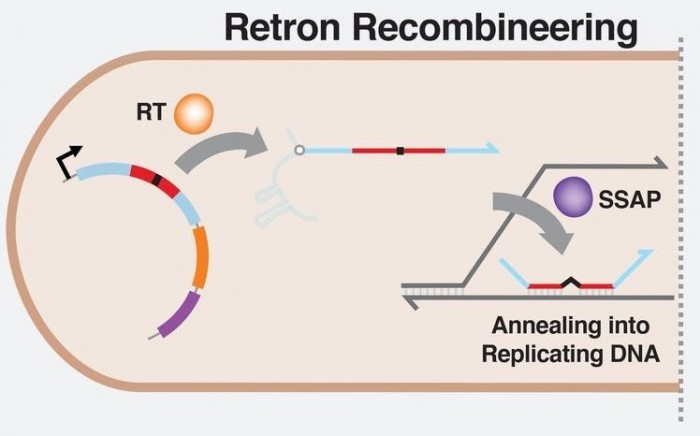
它是通过使用Retrons来实现的,Retrons是细菌DNA的片段,可以产生单链DNA片段。事实证明,这原本是一种自我防御机制,细菌用它来检查它们是否被病毒感染。通过添加所需的DNA片段和单链退火蛋白(SSAP),RLR系统确保在原始细胞分裂后,预期的DNA片段最终出现在子细胞的基因组中。
该研究的共同第一作者丹尼尔-古德曼(Daniel Goodman)说:"我们想,RLR应该让我们有能力在我们想要编辑的细胞内产生ssDNA,而不是试图从外部强迫它们进入细胞,而且不会破坏本地DNA,这都是非常引人注目的品质。"
这个新系统还有其他一些优势。它的扩展性很好,允许一次产生数百万个突变,而且随着细胞的复制,被编辑的细胞比例实际上也在不断增加。Retron序列也可以像商品"条形码"一样被追踪,让科学家在试图研究其效果时,可以轻松地检查哪些细胞接受了哪种编辑。
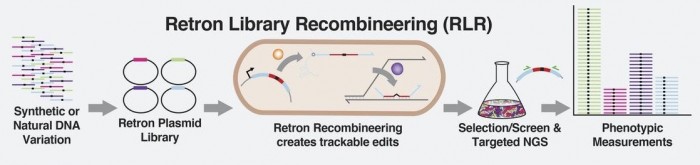
Retron Library Recombineering(RLR)可以加快关于细菌基因突变的实验室实验Max Schubert / 哈佛大学Wyss研究所
为了测试该系统,研究人员将其用于编辑大肠杆菌的种群。他们使用转录器向细菌引入抗生素抗性基因,并在对细菌进行了一些其他调整以阻止它们修复DNA "错误"后,他们发现超过90%的种群在20代后加入了所需的序列。由于转基因的条形码性质,研究小组能够轻松地跟踪哪些编辑将所需基因转移到细菌基因组中。
虽然还有很多工作要做,但该团队表示,新的RLR工具可以有一系列的应用。从短期来看,它可以成为研究细菌基因组和突变的一个强大的新工具,可能有助于创造新的有益菌株或发现诸如抗生素抗性等问题的治疗方案。从长远来看,它可能导致在其他生物体,甚至是人类中形成一种更安全的CRISPR替代品。
"该研究的高级作者George Church说:"能够用RLR分析集合的、条形码的突变体库,就能同时进行数百万次实验,使我们能够观察整个基因组的突变效果,以及这些突变可能如何相互作用。"这项工作有助于建立一个在其他遗传系统中使用RLR的路线图,这为未来的遗传研究提供了许多令人兴奋的可能性。
该研究发表在PNAS杂志上。